A Folding@Home está reunindo pesquisadores ao redor do mundo para entender melhor o Coronavírus 2019 (2019-nCoV), acelerando o esforço científico para desenvolver novas terapias e salvar vidas.
Parece complicado, mas não é. Através de simulações computacionais é possível aprimorar o conhecimento sobre Corona Vírus e testar possíveis medicamentos que possam ser usados no combate a COVID19. Os dados gerados pelo projeto serão liberados rapidamente de maneira aberta e divulgados como parte de uma colaboração científica ao redor do mundo, oferecendo aos pesquisadores novas ferramentas que podem abrir novas oportunidades para o desenvolvimento de medicamentos que salvam vidas.
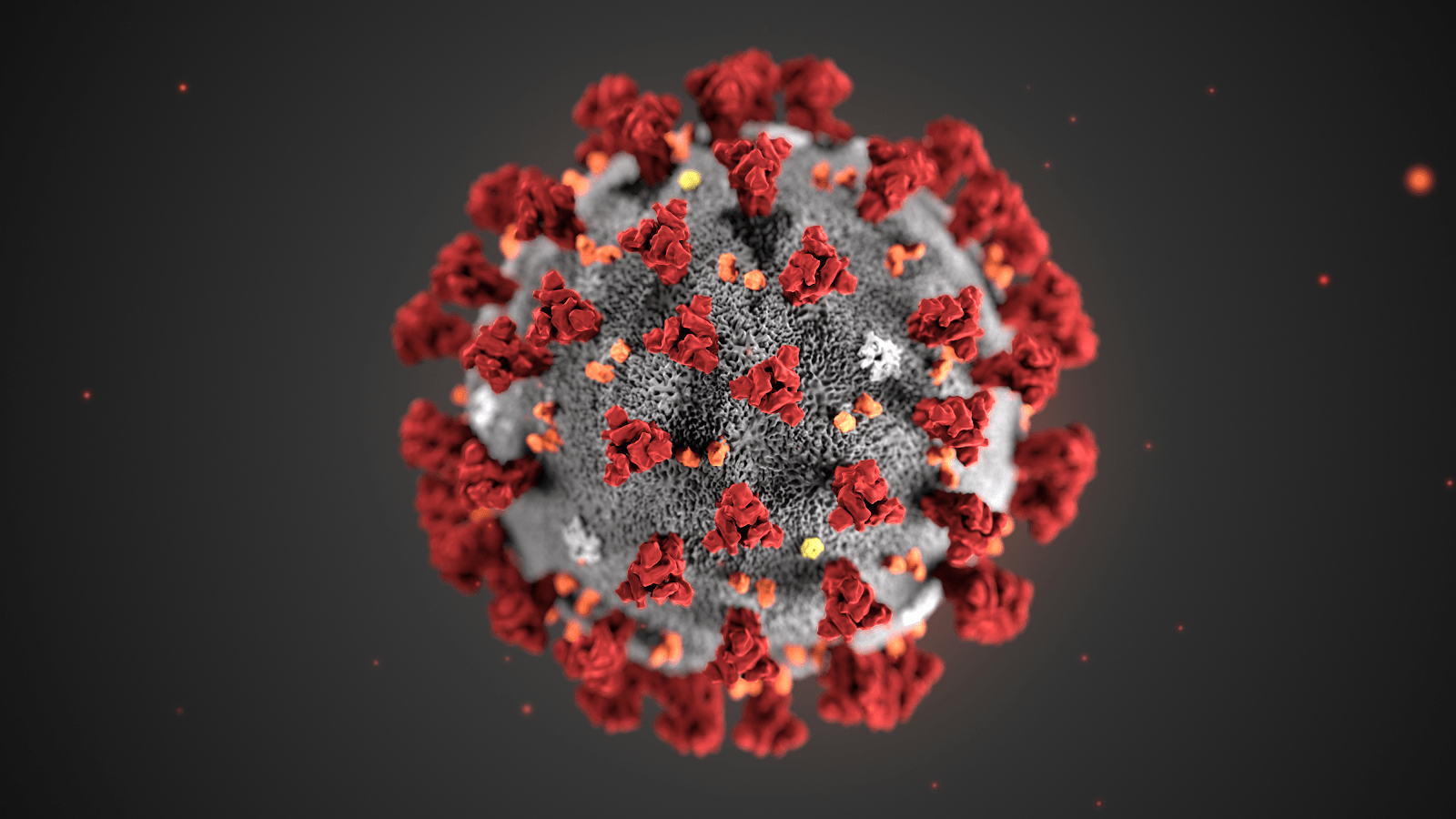
O 2019-nCoV é primo próximo do coronavírus SARS (SARS-CoV) e atua de maneira semelhante. Para ambos os coronavírus, a primeira etapa da infecção ocorre nos pulmões, quando uma proteína na superfície do vírus se liga a uma proteína receptora em uma célula do pulmão. Essa proteína viral é chamada de proteína spike, representada em vermelho na imagem abaixo, e o receptor é conhecido como ACE2. Um anticorpo terapêutico é um tipo de proteína que pode impedir a ligação da proteína viral ao seu receptor, impedindo, portanto, que o vírus infecte a célula pulmonar. Um anticorpo terapêutico já foi desenvolvido para SARS-CoV, mas para desenvolver anticorpos terapêuticos ou pequenas moléculas para 2019-nCoV, os cientistas precisam entender melhor a estrutura da proteína de pico viral e como ela se liga ao receptor ACE2 humano necessário para a entrada viral em células humanas.
As proteínas não são estagnadas – elas se mexem, se dobram e se desdobram para assumir várias formas. Precisamos estudar não apenas uma forma da proteína de pico viral, mas todas as maneiras pelas quais a proteína se mexe e se dobra em formas alternativas para entender melhor como ela interage com o receptor ACE2, para que um anticorpo possa ser projetado. Existem estruturas de baixa resolução da proteína spike SARS-CoV e conhecemos as mutações que diferem entre SARS-CoV e 2019-nCoV. Dada essa informação, estamos posicionados de maneira exclusiva para ajudar a modelar a estrutura da proteína spike 2019-nCoV e identificar locais que podem ser direcionados para um anticorpo terapêutico. Podemos construir modelos computacionais que atingem esse objetivo, mas é preciso muito poder de computação.
É aqui que você entra! Com muitos computadores trabalhando para o mesmo objetivo, nosso objetivo é ajudar a desenvolver um remédio terapêutico o mais rápido possível.
Passo a passo para colaborar
Faça o download do Folding@home para windows clicando aqui: fah-installer_7.5.1_x86.exe
Execute e siga os passos da instalação.
Seu navegador padrão vai abrir com essa imagem:
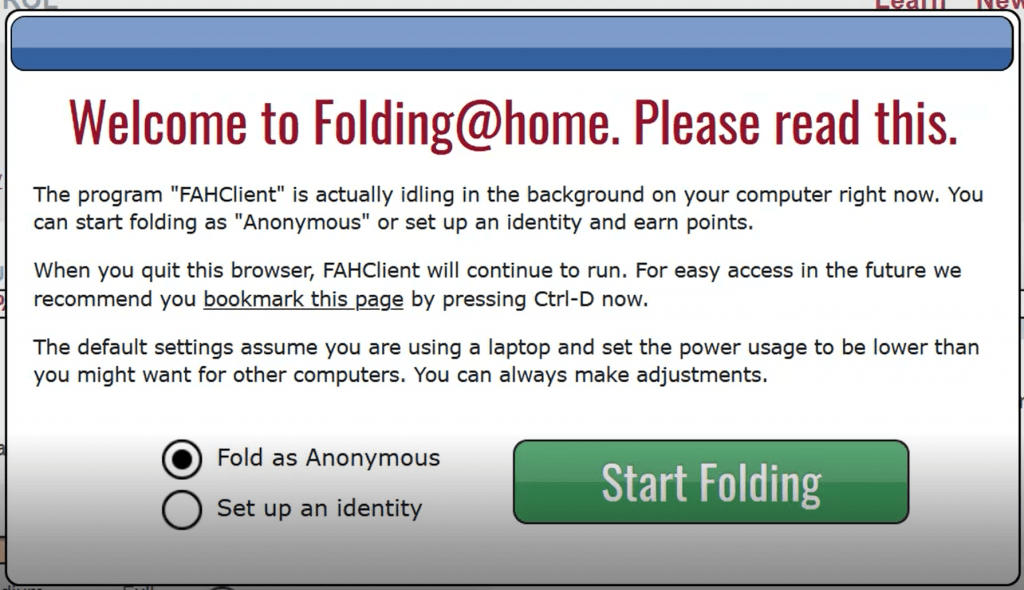
Você pode fazer de maneira anônima ou pode participar da equipe de Cascavel, e saber quantas simulações você e a equipe conseguiram realizar.
Para participar da equipe, você deve selecionar a opção “Set up an identity” e clicar em Start Folding
Para criar seu usuário entre nesse link aqui: https://apps.foldingathome.org/getpasskey
Vai aparecer a seguinte tela:
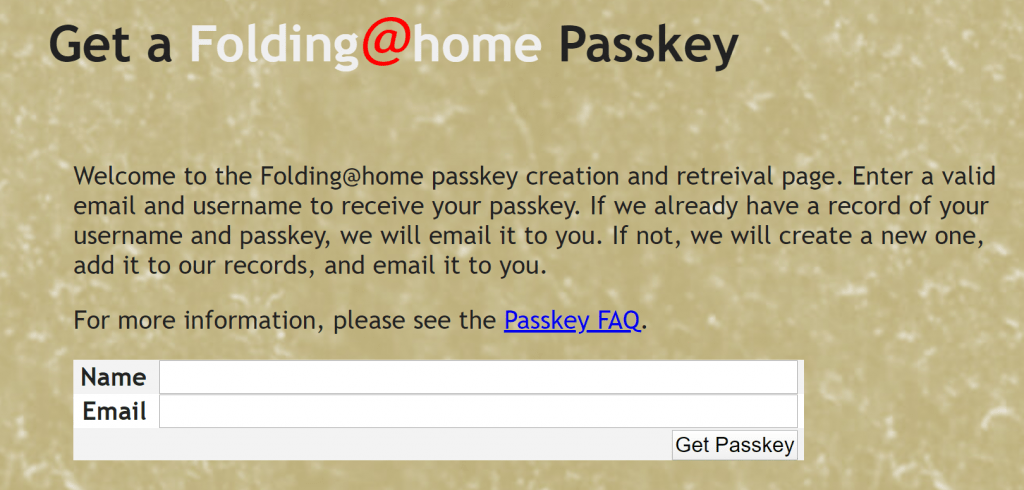
Digite seu nome e email, e depois clique em: “Get Passkey”
Acesse o seu e-mail onde receberá as informações de “Username” e “Passkey” que deverá copiar e colar na tela seguinte:
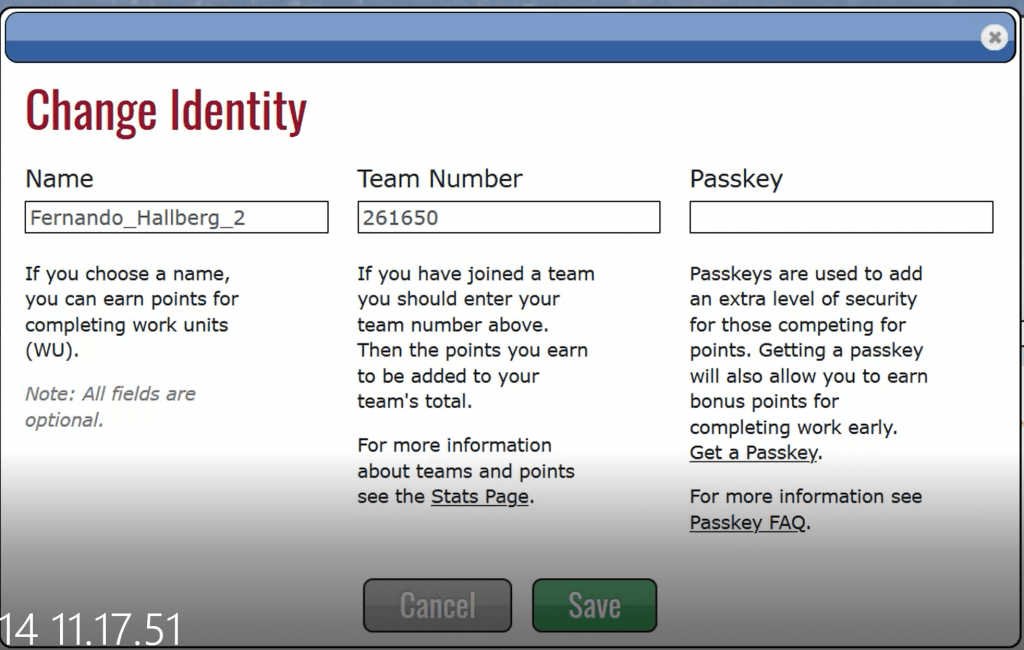
Onde está escrito “Name”, coloque o nome de usuário que recebeu no e-mail.
Onde está escrito “Team Number”, digite 261650 – que é o número da equipe COVID19-Cascavel
Onde está escrito “Passkey”, coloque a chave que recebeu junto com o nome do usuário no seu e-mail!
Qualquer dúvida pode enviar via chat aqui pelo site, ou por nossa página no facebook: http://fb.com/fernandohallberg.oficial






